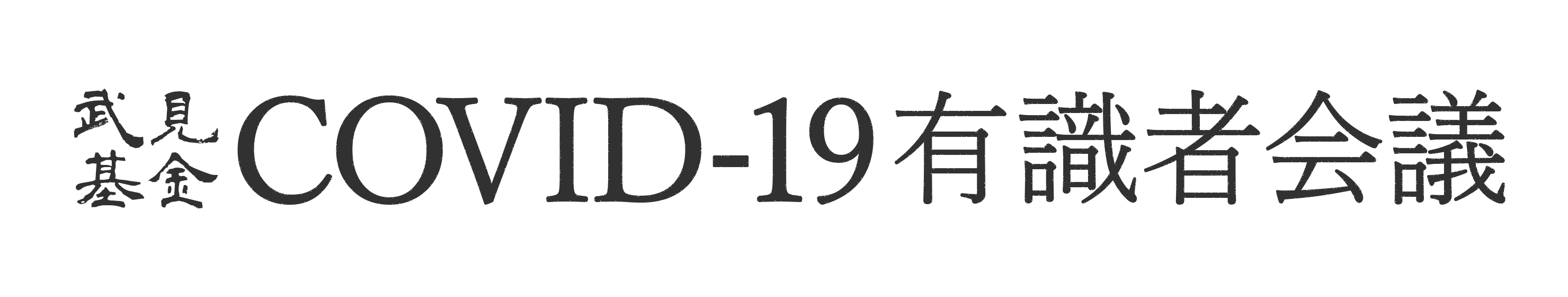- コロナウイルスは、ウイルスゲノムとして一本鎖のプラス鎖RNAを持つエンベロープウイルスである。表面上に特徴のある王冠のような突起(スパイク)を有するため、ラテン語の王冠を意味する”corona”という名前が付けられた。
- コロナウイルスの宿主域は広く、本ウイルスは、ヒトや野生動物だけでなく、家畜、愛玩動物、実験動物など多くの動物に感染し、様々な疾患を起こす。
- 系統進化学的な解析によって、ヒトに感染する既知のコロナウイルスは全て、野生動物であるコウモリやげっ歯類に由来することが示唆されている。
- SARS-CoV-2の自然宿主もコウモリである可能性が高い。SARS-CoV-2は、SARS-CoV-1と同様に、ヒトのACE2と結合することによって、宿主細胞に侵入することが報告されている。
- アカゲザル、ハムスター、フェレットはSARS-CoV-2に対する感受性が高いので、SARS-CoV-2の病原性解析および治療薬やワクチンの評価試験を行うための良い動物モデルになると考えられる。
コロナウイルスの基本性状
コロナウイルスの構造
コロナウイルスは、ウイルスゲノムとして一本鎖のプラス鎖RNAを持つエンベロープウイルスである。ウイルスは直径約100nmの球形を示しており、表面上に特徴のある王冠のような突起(スパイク)を有するため、ラテン語の王冠を意味する”corona”という名前が付けられた【図表1】。
コロナウイルスのゲノムサイズは、約27〜32キロベース (kb)であり、現在知られているRNAウイルスの中では最大である。SARS-CoV-2のゲノムRNAのサイズは、約30kbである。コロナウイルスのゲノムRNAは、5’末端にキャップ構造、3’末端にポリA配列を有しており、宿主細胞に感染して、そのままmRNAとして機能することができる。ウイルスRNAの5’末端側の約20kbには2つのオープンリーデイングフレーム(ORF1aおよびORF1b)があり、ORF1aの終止コドンの少し上流にORF1bの開始コドンが存在する。ORF1aとORF1bからは、1a、および、リボソームのフレームシフトによって合成される1a+1bという2つの蛋白質が翻訳される【図表1】。これらの蛋白質は自らが持つプロテアーゼによって、RNA依存性RNAポリメラーゼなどを含む十数種類の非構造蛋白質に開裂する。
ウイルスRNAの下流の約10 kbは、構造蛋白質であるS(スパイク)蛋白質、E(エンベロープ)蛋白質、M(メンブラン)蛋白質、N(ヌクレオカプシド)蛋白質をコードしている。N蛋白質は、ウイルスゲノムRNAと結合してヌクレオカプシドを形成する。ヌクレオカプシドがエンベロープによって包み込まれることによって、ウイルス粒子が形成される。エンベロープには、S蛋白質、E蛋白質、M蛋白質が配置されている【図表1】。S蛋白質は、ウイルスが宿主細胞に侵入する際(感染初期)に重要な役割を果たし、E蛋白質とM蛋白質はウイルスアセンブリーと出芽(感染後期)に必要である。
| 図表1 |
| コロナウイルスの構造 |
| コロナウイルスは、ウイルスゲノムとして一本鎖のプラス鎖RNAを持つ。ウイルス粒子内部には、N蛋白質とウイルスゲノムRNAが結合して形成するヌクレオカプシドが存在し、エンベロープで包み込まれている。エンベロープには、S蛋白質、E蛋白質、M蛋白質が配置されている。 |
 |
コロナウイルスの生活環
コロナウイルスの宿主細胞への感染は、エンベロープ上のS蛋白質が、細胞表面に発現する受容体(レセプター)と結合することから始まる。S蛋白質は、宿主細胞のプロテアーゼによって、S1とS2という2つのサブユニットに開裂される。S1はレセプター結合、そしてS2はウイルスエンベロープと細胞との膜融合において、役割を担う。S1に存在するレセプター結合部位(receptor binding domain; RBD)が、細胞表面のレセプターと結合する。
S蛋白質のレセプター特異性は、コロナウイルスの宿主域や組織向性(特定の組織や臓器に選択的に感染する性質)を決定する主要な要因である。SARS-CoV-1のレセプターとしてAngiotensin converting enzyme-1 (ACE2)が、MERS-CoVのレセプターとしてDipeptidyl peptidase 4 (DPP4)、HCoV-229EのレセプターとしてAminopeptidase N (APN)、そしてHCoV-OC43とHCoV-HKU1のレセプターとして9-O-acetylated sialic acidが、それぞれ同定されている [1]。
コロナウイルスの細胞侵入機構としては、レセプターへの結合後、1)細胞表面から細胞内へ侵入する経路と、2)エンドサイトーシスによって、ウイルス粒子が細胞内に取り込まれた後、エンドソームから細胞内へと侵入する経路がある。エンベロープウイルスが細胞に侵入する際、ウイルスエンベロープと細胞の脂質二重膜とが融合する必要がある。コロナウイルスの場合、S蛋白質のサブユニットS2はFusion Peptideを有しており、膜融合において重要な役割を果たす。SARS-CoV-1では、上記2)が主な経路と考えられており、エンドサイトーシスによって取り込まれたウイルスのS蛋白質が、宿主プロテアーゼによって活性化され、エンドソームとウイルスエンベロープとの膜融合を起こす。SARS-CoV-1のS蛋白質を活性化(S1とS2に開裂)しうる宿主プロテアーゼとしては、カテプシン、トリプシン、エラスターゼ、TMPRSS2が報告されている[2–5]。またMERS-CoVのS蛋白質はFurinによってS1とS2に開裂される[6]。
膜融合後、ウイルスの脱殻が起こり、ウイルスゲノムが細胞内に放出されたのち、細胞質内でウイルス複製が始まる。コロナウイルスのプラス鎖ゲノムRNAは、そのままmRNAとして機能できるため、宿主細胞のリボソームに結合して、ウイルス複製に必要なRNA依存性RNAポリメラーゼなどが合成される。プラス鎖ゲノムRNAを鋳型として、合成された相補的マイナス鎖RNAをもとにして、それぞれのウイルス蛋白質をコードするmRNAが転写され、ウイルス蛋白質が作られる。また子孫ウイルス用のプラス鎖ゲノムRNAの複製も行われる。
新たに合成されたウイルス構造蛋白質(S、E、M蛋白質)は、小胞体―ゴルジ装置中間体(ERGIC)に輸送される。N蛋白質とウイルスRNAによって形成されたヌクレオカプシドは、他の構造蛋白質とともに、ウイルス粒子を形成し、ERGIC内へ出芽する。M蛋白質とE蛋白質が、ウイルスの出芽ステップにおいて重要な役割を果たす。ERGIC内に出芽した子孫ウイルスは、エクソサイトーシスによって、細胞外へと放出される。
SARS-CoV-2のウイルス学的特徴について
SARS-Cov-2の分類と起源
鳥類や哺乳類に感染するコロナウイルスは、ニドウイルス目コロナウイルス科オルソコロナウイルス亜科に属する。オルソコロナウイルス亜科には、アルファコロナウイルス属、ベータコロナウイルス属、ガンマコロナウイルス属、デルタコロナウイルス属という4つの属が含まれる【図表2】。
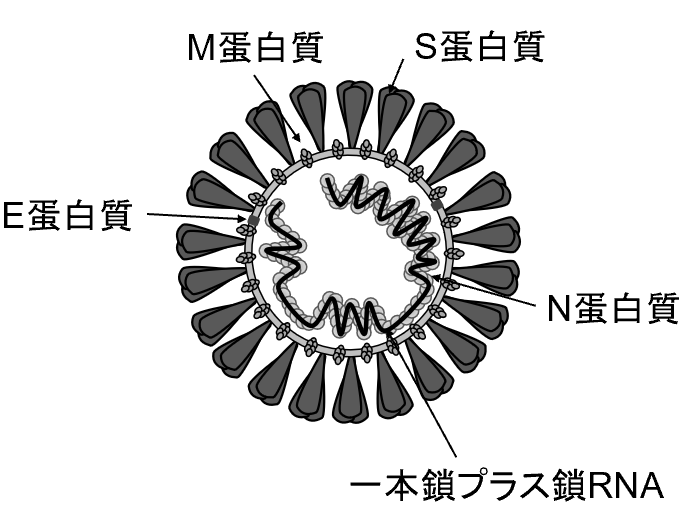
ヒトに感染するコロナウイルスとして、現在、7種類のウイルスが知られている。ヒトに日常的に感染するヒトコロナウイルス(Human coronavirus; HCoV)であるHCoV-229E、HCoV-NL63、HCoV-OC43、HCoV-HKU1と、2003年に重症急性呼吸器症候群(SARS)を引き起こしたSARSコロナウイルス (SARS-CoV-1)、2012年に出現した中東呼吸器症候群(MERS)コロナウイルス (MERS-CoV)、そして現在パンデミックを引き起こしている新型コロナウイルス (SARS-CoV-2)である【図表2】。
上記7種類のウイルスのうち、HCoV-229EとHCoV-NL63はアルファコロナウイルス属、そして残りの5種類のウイルス(HCoV-OC43、HCoV-HKU1、SARS-CoV-1、MERS-CoV、SARS-CoV-2)はベータコロナウイルス属に分類される。ベータコロナウイルス属は4つの系統(A, B, C, D系統)に分かれており、SARS-CoV-1とSARS-CoV-2はB系統に属する【図表2】。
| 図表2 |
| SARS-CoV-2の分類 |
| SARS-CoV-1、MERS-CoV、SARS-CoV-2はベータコロナウイルス属に分類される。ベータコロナウイルス属は4つの系統(A, B, C, D系統)に分かれており、SARS-CoV-1とSARS-CoV-2はB系統に属する。2013年に中国の野生のコウモリから分離されたbat/Yunnan/RaTG13/2013、および、2019年にマレーセイザンコウから分離されたウイルスは、SARS-CoV-2と近縁である。 |
 |
系統進化学的な解析によって、上記のヒトに感染するコロナウイルスは全て、野生動物であるコウモリやげっ歯類に由来することが示唆されている[7,8]。もともとコウモリやげっ歯類などの自然宿主が保有するコロナウイルスが、中間宿主への感染を経て、最終的にヒトに感染し、病気を起こすようになったと考えられている。
SARS-CoV-2については、コウモリから、本ウイルスに近縁のコロナウイルスの配列が見つかっているため、SARS-CoV-2の自然宿主もコウモリである可能性が高い[9–11]。また、マレーセンザンコウからもSARS-CoV-2に近縁のコロナウイルスが検出されているため、マレーセンザンコウが中間宿主であるという説があるが、詳細は不明である[12,13]。今後のさらなる解析によって、SARS-CoV-2の自然宿主および中間宿主(もし存在すれば)が明らかとなることが期待される。
SARS-CoV-2のレセプター
SARS-CoV-2のゲノムRNAやウイルス蛋白質における、SARS-CoV-1との相同性は、ゲノムRNAが79.0%、S蛋白質が76.2%、E蛋白質が94.7%、M蛋白質が90.1%、N蛋白質が90.3%である[11]。X線結晶構造解析やクライオ電子顕微鏡による単粒子構造解析などによって、S蛋白質(および他のウイルス蛋白質)の構造解析が進んでいる[14,15]。
SARS-CoV-2のS蛋白質は、SARS-CoV-1と同様に、ヒトのACE2と結合することによって、宿主細胞に侵入することが、いくつかの研究グループによって報告されている[10,14,16–18]。また、SARS-CoV-2のS蛋白質は、MERS-CoVのレセプターであるDPP4とHCoV-229EのレセプターであるAPNは認識しないことも示されている[10,16–18]。SARS-CoV-2のS蛋白質のRBDとACE2とが結合して形成された複合体の構造も明らかにされている[19]。
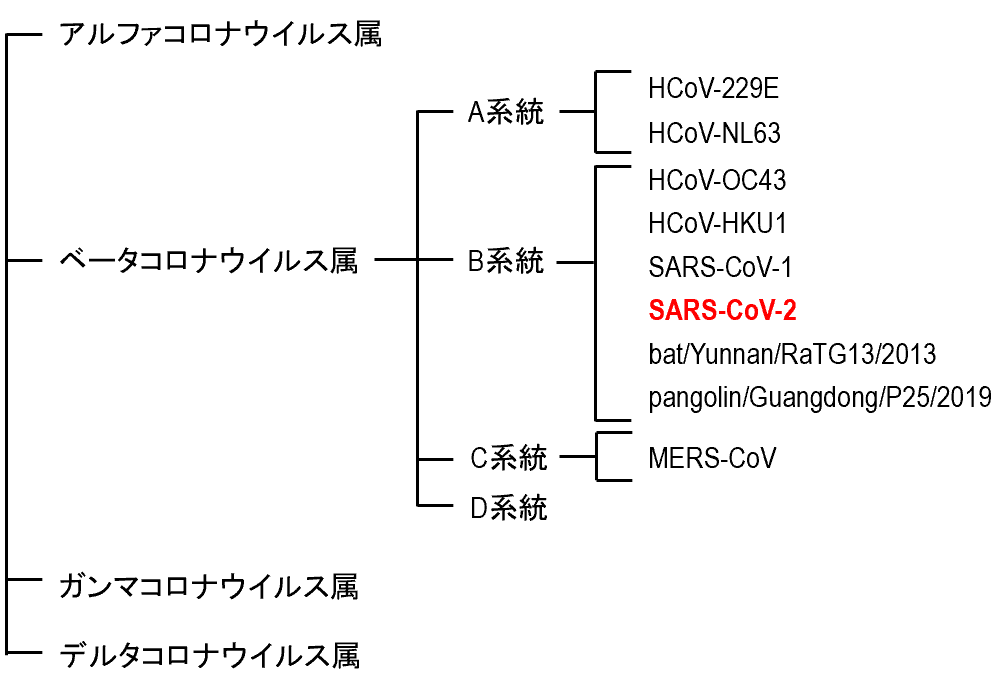
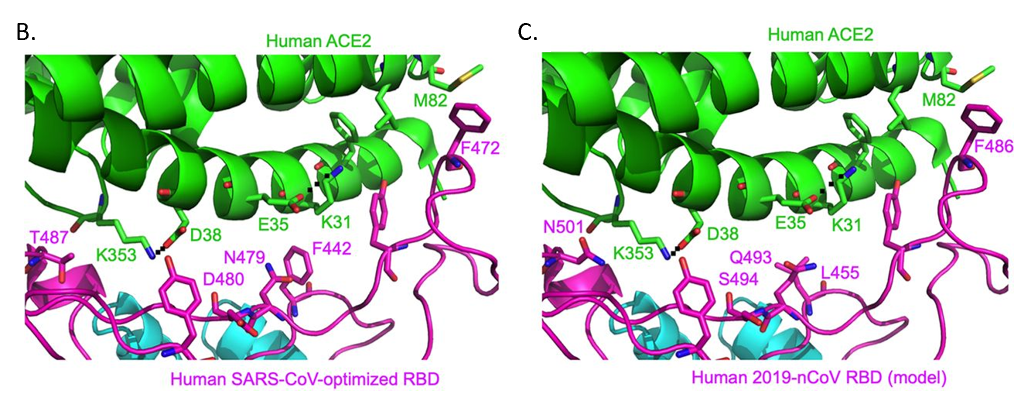
SARS-CoV-1のS蛋白質のRBDは、コア構造とレセプター結合モチーフ(RBM)によって構成されており、RBMはACE2表面と直接結合する[20] 【図表3A】。SARS-CoV-1の研究により、RBMのY442, L472, N479, D480, T487,Y491の6つのアミノ酸が、ACE2との結合に重要であること、および、SARS関連コロナウイルス の宿主域の決定に関わることが示されている[21]。SARS-CoV-2において、これら6つのアミノ酸に対応するものはL455, F486, Q493, S494, N501, Y505であるが、Y505(SARS-CoV-1ではY491)以外は、SARS-CoV-1のアミノ酸とは異なっている【図表3B、C】。蛋白質構造のコンピューター解析によって、SARS-CoV-2のRBMと、ヒトを含む様々な動物種のACE2との結合親和性を調べたところ、SARS-CoV-2のRBMは、ヒト、ジャコウネズミ、ブタ、フェレット、ネコ、オランウータン、サル(ミドリザル)、コウモリ(キクガシラコウモリ科)のACE2と結合親和性が高いこと、および、マウスとラットのACE2との結合親和性は低いことが予測された[21]。したがって、SARS-CoV-2は上記の動物種(マウスとラットを除く)に感染する可能性があると考えられる。
| 図表3 |
| SARS-CoV-1およびSARS-CoV-2のS蛋白質のレセプター結合部位とヒトACE2 |
| SARS-CoV-1のS蛋白質のRBDは、コア構造とレセプター結合モチーフ(RBM)によって構成されており、RBMはACE2表面と直接結合する(A)。 SARS-CoV-1(B)およびSARS-CoV-2(C)のS蛋白質のレセプター結合モチーフとヒトACE2との結合部位を示す(SARS-CoV-2はモデル)。Yushun Wan et al. J. Virol. 2020; doi:10.1128/JVI.00127-20より引用 |
  |
S蛋白質のFurin切断サイト
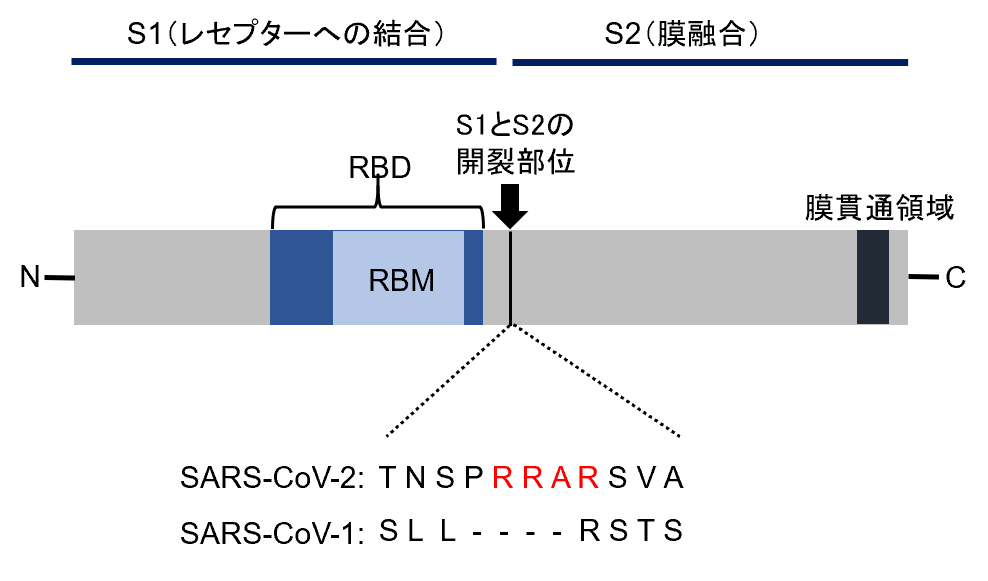
SARS-CoV-2とSARS-CoV-1 との大きな違いの一つに、SARS-CoV-2のS蛋白質のS1/S2開裂部位に、塩基性アミノ酸が連続した特徴的な配列(RRAR)を持つことが挙げられる。この配列は“Furin切断サイト”と呼ばれ、Furinや他のプロテアーゼによって効率よく切断される【図表4】。このFurin切断サイトは、SARS-CoV-1にはないが、MERS-CoV、およびHCoV-OC43のS蛋白質には存在する[14,22]。
| 図表4 |
| S蛋白質の構造 |
| SARS-CoV-2のS蛋白質には、S1/S2開裂部位に、塩基性アミノ酸が連続した特徴的な配列(RRAR)が存在し、この配列は“Furin切断サイト”と呼ばれる。この配列は、SARS-CoV-1にはない。 |
 |
“コロナウイルス の生活環”の項で述べたように、ウイルス増殖サイクルにおいて、S蛋白質はS1とS2に開裂するが、コロナウイルスの種類によって、開裂を受ける場所やタイミングが異なる。すなわち、1)感染細胞において、S蛋白質が合成されたのち、宿主プロテアーゼによって開裂する場合と、2)ウイルスが標的細胞に侵入する際に、S蛋白質がレセプターに結合した後、宿主プロテアーゼによって開裂する場合である。SARS-CoV-1での仕組みは後者であるため、ウイルス粒子表面にはS蛋白質が非開裂の状態で存在しており、ウイルスが細胞に侵入する際に宿主プロテアーゼ(トリプシン、エラスターゼ、カテプシン、TMPRSS2)によって開裂する。それに対して、SARS-CoV-2の場合は、1)の仕組みによって、S蛋白質合成後に細胞内で開裂が起こる。シュードタイプウイルスを用いた実験において、ウイルス粒子のS蛋白質が、開裂型のS1とS2として存在することが示されている[14,16,17]。またシュードタイプウイルスを用いた実験によって、SARS-CoV-2が効率良くヒトの呼吸器に感染するためには、S蛋白質のFurin切断サイトが必要であること、および、TMPRSS2によるS蛋白質の活性化が重要であることが示唆されている[16,23]。
様々な動物モデルを用いたウイルスの性状解析
コロナウイルスの宿主域は広く、本ウイルスは、ヒトや野生動物だけでなく、家畜、愛玩動物、実験動物など多くの動物に感染し、様々な疾患を起こす。SARS-CoV-2の場合、香港においてペットのイヌ2匹、およびネコ1匹から本ウイルスが検出されており、また、米国ニューヨーク州においては、ペットのネコ2匹、および動物園のトラやライオンなどのネコ科動物で本ウイルスの感染が報告されている。
国内外の研究室において、様々な実験動物を用いたSARS-CoV-2の性状解析が行われている。中国のグループが、人間との距離が近い家畜やペットなどの動物種について、SARS-CoV-2に対する感受性を調べたところ、フェレットとネコに呼吸器では本ウイルスが増殖したが、イヌ、ブタ、ニワトリ、アヒルではウイルスは増殖しないことが分かった[24] 。また韓国のグループは、フェレットにおいて、SARS-CoV-2が飛沫伝播を起こすことを報告している[25]。
東京大学医科学研究所の河岡義裕教授の研究グループは、米国ウイスコンシン大学との共同研究によって、ネコにおけるSARS-CoV-2の増殖性と感染伝播性の解析を行い、本ウイルスが、ネコの呼吸器でよく増えること、および、接触感染によってネコの間で容易に感染伝播することを明らかにした[26] 【図表5】。また、本ウイルスに感染したネコは明らかな症状を示さなかった。本研究成果は、SARS-CoV-2がネコの間で広がる可能性があることを示唆している。
| 図表5 |
| ネコを用いたSARS-CoV-2感染伝播実験 |
| SARS-CoV-2をネコの鼻腔内に接種した。その後、感染ネコとの接触によってウイルスが同居ネコに伝播するのかどうかを調べたところ、3ペア中3ペアにおいてウイルスが伝播した。また、ウイルスはネコの呼吸器でよく増えることも分かった。 東京大学医科学研究所HPより転載 https://www.ims.u-tokyo.ac.jp/imsut/jp/about/press/page_00004.html |
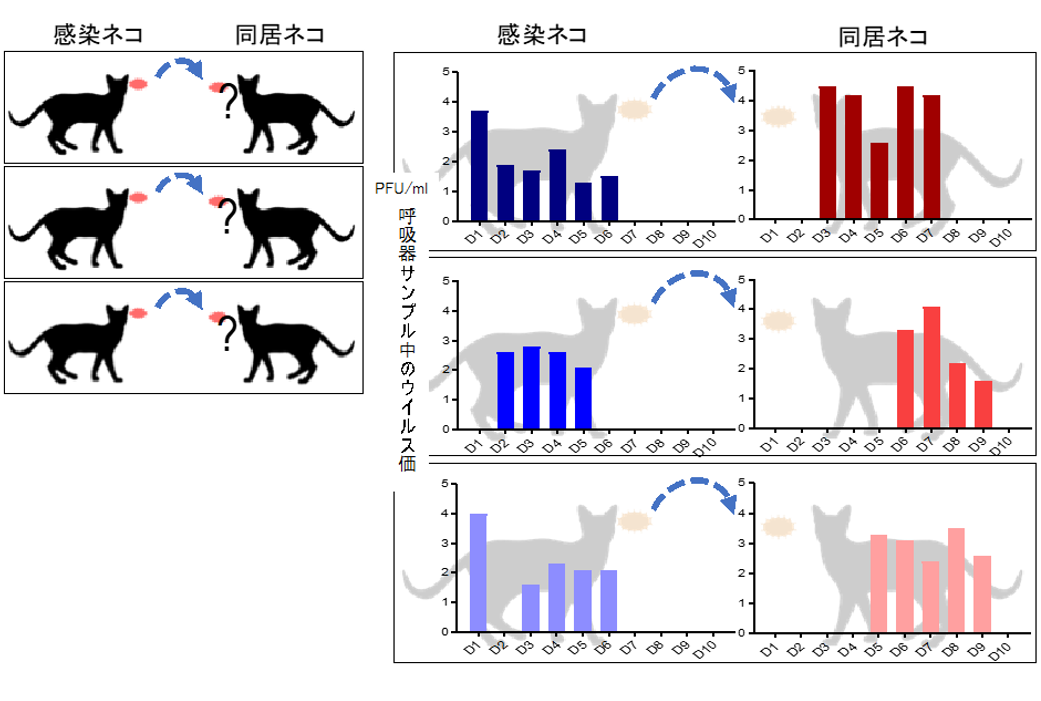 |
ハムスターモデルにおけるSARS-CoV-2の感染系も確立されている。本ウイルスがハムスターの呼吸器においてよく増殖すること、および、肺においてCOVID-19患者の肺炎像と類似の病変を引き起こすことが明らかとなっている。また、感染個体の回復期血清を投与した非感染のハムスターにおいては、SARS-CoV-2の増殖が阻害されることが示された[27]。さらに、ハムスター間で、接触あるいは飛沫によって本ウイルスの感染伝播が起こることが示されている[27,28]。他の齧歯類モデルとしては、ヒトのACE2を発現しているトランスジェニックマウス (hACE2マウス)が挙げられ、SARS-CoV-2を感染させたhACE2マウスの肺において、典型的な間質性肺炎の病理所見が観察された [29]。このような齧歯類モデルは、新型コロナに対する治療薬やワクチンの評価試験において、大いに有用である。
霊長類モデルとしては、アカゲザルが有望であることが報告されている[30,31]。SARS-CoV-2を感染させたアカゲザルは呼吸器症状を示し、その症状は8日〜16日程度継続した。胸部X線写真では、ヒトのCOVID-19患者に見られるような肺浸潤の様子が観察された。また3〜5歳のアカゲザルと15歳のアカゲザルのグループを比べたところ、高齢のサルの方が、より重度なびまん性間質性肺炎を起こしていた[30]。この結果は、高齢者のCOVID-19感染者が重症化しやすいという現象を反映していることから、アカゲザルはSARS-CoV-2の病原性解析における良いモデルになると考えられる。
[引用文献]
- Fung, T.S.; Liu, D.X. Human Coronavirus: Host-Pathogen Interaction. Annu. Rev. Microbiol. 2019, 73, 529–557.
- Simmons, G.; Gosalia, D.N.; Rennekamp, A.J.; Reeves, J.D.; Diamond, S.L.; Bates, P. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry. Proc. Natl. Acad. Sci. U. S. A. 2005, 102, 11876–11881.
- Simmons, G.; Reeves, J.D.; Rennekamp, A.J.; Amberg, S.M.; Piefer, A.J.; Bates, P. Characterization of severe acute respiratory syndrome-associated coronavirus (SARS-CoV) spike glycoprotein-mediated viral entry. Proc. Natl. Acad. Sci. U. S. A. 2004, 101, 4240–4245.
- Matsuyama, S.; Nagata, N.; Shirato, K.; Kawase, M.; Takeda, M.; Taguchi, F. Efficient Activation of the Severe Acute Respiratory Syndrome Coronavirus Spike Protein by the Transmembrane Protease TMPRSS2. J. Virol. 2010, 84, 12658–12664.
- Matsuyama, S.; Ujike, M.; Morikawa, S.; Tashiro, M.; Taguchi, F. Protease-mediated enhancement of severe acute respiratory syndrome coronavirus infection. Proc. Natl. Acad. Sci. U. S. A. 2005, 102, 12543–12547.
- Millet, J.K.; Whittaker, G.R. Host cell proteases: Critical determinants of coronavirus tropism and pathogenesis. Virus Res. 2015, 202, 120–134.
- Forni, D.; Cagliani, R.; Clerici, M.; Sironi, M. Molecular Evolution of Human Coronavirus Genomes. Trends Microbiol. 2017, 25, 35–48.
- Cui, J.; Li, F.; Shi, Z.L. Origin and evolution of pathogenic coronaviruses. Nat. Rev. Microbiol. 2019, 17, 181–192.
- Wu, F.; Zhao, S.; Yu, B.; Chen, Y.M.; Wang, W.; Song, Z.G.; Hu, Y.; Tao, Z.W.; Tian, J.H.; Pei, Y.Y.; et al. A new coronavirus associated with human respiratory disease in China. Nature 2020, 579, 265–269.
- Zhou, P.; Yang, X. Lou; Wang, X.G.; Hu, B.; Zhang, L.; Zhang, W.; Si, H.R.; Zhu, Y.; Li, B.; Huang, C.L.; et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature 2020, 579, 270–273.
- Lu, R.; Zhao, X.; Li, J.; Niu, P.; Yang, B.; Wu, H.; Wang, W.; Song, H.; Huang, B.; Zhu, N.; et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet 2020, 395, 565–574.
- Lam, T.T.Y.; Shum, M.H.H.; Zhu, H.C.; Tong, Y.G.; Ni, X.B.; Liao, Y.S.; Wei, W.; Cheung, W.Y.M.; Li, W.J.; Li, L.F.; et al. Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins. Nature 2020.
- Xiao, K.; Zhai, J.; Feng, Y.; Zhou, N.; Zhang, X.; Zou, J.-J.; Li, N.; Guo, Y.; Li, X.; Shen, X.; et al. Isolation of SARS-CoV-2-related coronavirus from Malayan pangolins. Nature 2020.
- Walls, A.C.; Park, Y.J.; Tortorici, M.A.; Wall, A.; McGuire, A.T.; Veesler, D. Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein. Cell 2020, 181, 281-292.e6.
- Wrapp, D.; Wang, N.; Corbett, K.S.; Goldsmith, J.A.; Hsieh, C.L.; Abiona, O.; Graham, B.S.; McLellan, J.S. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science (80-. ). 2020, 367, 1260–1263.
- Hoffmann, M.; Kleine-Weber, H.; Schroeder, S.; Krüger, N.; Herrler, T.; Erichsen, S.; Schiergens, T.S.; Herrler, G.; Wu, N.H.; Nitsche, A.; et al. SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor. Cell 2020, 181, 271-280.e8.
- Ou, X.; Liu, Y.; Lei, X.; Li, P.; Mi, D.; Ren, L.; Guo, L.; Guo, R.; Chen, T.; Hu, J.; et al. Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV. Nat. Commun. 2020, 11.
- Letko, M.; Marzi, A.; Munster, V. Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses. Nat. Microbiol. 2020, 5, 562–569.
- Yan, R.; Zhang, Y.; Li, Y.; Xia, L.; Guo, Y.; Zhou, Q. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science (80-. ). 2020, 367, 1444–1448.
- Li, F.; Li, W.; Farzan, M.; Harrison, S.C. Structural biology: Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science (80-. ). 2005, 309, 1864–1868.
- Wan, Y.; Shang, J.; Graham, R.; Baric, R.S.; Li, F. Receptor Recognition by the Novel Coronavirus from Wuhan: an Analysis Based on Decade-Long Structural Studies of SARS Coronavirus. J. Virol. 2020, 94, 1–9.
- Coutard, B.; Valle, C.; de Lamballerie, X.; Canard, B.; Seidah, N.G.; Decroly, E. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 2020, 176.
- Hoffmann, M.; Kleine-Weber, H.; Pöhlmann, S. A Multibasic Cleavage Site in the Spike Protein of SARS-CoV-2 Is Essential for Infection of Human Lung Cells. Mol. Cell 2020.
- Shi, J.; Wen, Z.; Zhong, G.; Yang, H.; Wang, C.; Huang, B.; Liu, R.; He, X.; Shuai, L.; Sun, Z.; et al. Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS–coronavirus 2. Science (80-. ). 2020, eabb7015.
- Kim, Y. Il; Kim, S.G.; Kim, S.M.; Kim, E.H.; Park, S.J.; Yu, K.M.; Chang, J.H.; Kim, E.J.; Lee, S.; Casel, M.A.B.; et al. Infection and Rapid Transmission of SARS-CoV-2 in Ferrets. Cell Host Microbe 2020, 1–6.
- Halfmann, P.J.; Hatta, M.; Chiba, S.; Maemura, T.; Fan, S.; Takeda, M.; Kinoshita, N.; Hattori, S.; Sakai-Tagawa, Y.; Iwatsuki-Horimoto, K.; et al. Transmission of SARS-CoV-2 in Domestic Cats. N. Engl. J. Med. 2020.
- Chan, J.F.-W.; Zhang, A.J.; Yuan, S.; Poon, V.K.-M.; Chan, C.C.-S.; Lee, A.C.-Y.; Chan, W.-M.; Fan, Z.; Tsoi, H.-W.; Wen, L.; et al. Simulation of the clinical and pathological manifestations of Coronavirus Disease 2019 (COVID-19) in golden Syrian hamster model: implications for disease pathogenesis and transmissibility. Clin. Infect. Dis. 2020.
- Sia, S.F.; Yan, L.-M.; Chin, A.W.; Fung, K.; Poon, L.L.; Nicholls, J.M.; School, M.P.; Yen, H.-L. Pathogenesis and transmission of SARS-CoV-2 virus in golden Syrian hamsters. Nat. Res. Rev. 2020, 1–16.
- Bao L. et al. The Pathogenicity of 2019 Novel Coronavirus in hACE2 Transgenic Mice. bioRxiv 2020, 1–13.
- Yu, P.; Qi, F.; Xu, Y.; Li, F.; Liu, P.; Liu, J.; Bao, L.; Deng, W.; Gao, H.; Xiang, Z.; et al. Age‐related rhesus macaque models of COVID‐19. Anim. Model. Exp. Med. 2020, 3, 93–97.
- Munster, V.; Feldmann, F.; Williamson, B.; Doremalen, N. van; Lizzette Perez-Perez; Schultz, J.; Meade-White, K.; Okumura, A.; Callison, J.; Brumbaugh, B.; et al. Respiratory disease and virus shedding in rhesus macaques inoculated with SARS-CoV-2. bioRxiv 2020, 2020.03.21.001628.